forked from DU-Bii/module-5-Methodes-Outils
-
Notifications
You must be signed in to change notification settings - Fork 0
/
slides.html
935 lines (735 loc) · 31.7 KB
/
slides.html
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
50
51
52
53
54
55
56
57
58
59
60
61
62
63
64
65
66
67
68
69
70
71
72
73
74
75
76
77
78
79
80
81
82
83
84
85
86
87
88
89
90
91
92
93
94
95
96
97
98
99
100
101
102
103
104
105
106
107
108
109
110
111
112
113
114
115
116
117
118
119
120
121
122
123
124
125
126
127
128
129
130
131
132
133
134
135
136
137
138
139
140
141
142
143
144
145
146
147
148
149
150
151
152
153
154
155
156
157
158
159
160
161
162
163
164
165
166
167
168
169
170
171
172
173
174
175
176
177
178
179
180
181
182
183
184
185
186
187
188
189
190
191
192
193
194
195
196
197
198
199
200
201
202
203
204
205
206
207
208
209
210
211
212
213
214
215
216
217
218
219
220
221
222
223
224
225
226
227
228
229
230
231
232
233
234
235
236
237
238
239
240
241
242
243
244
245
246
247
248
249
250
251
252
253
254
255
256
257
258
259
260
261
262
263
264
265
266
267
268
269
270
271
272
273
274
275
276
277
278
279
280
281
282
283
284
285
286
287
288
289
290
291
292
293
294
295
296
297
298
299
300
301
302
303
304
305
306
307
308
309
310
311
312
313
314
315
316
317
318
319
320
321
322
323
324
325
326
327
328
329
330
331
332
333
334
335
336
337
338
339
340
341
342
343
344
345
346
347
348
349
350
351
352
353
354
355
356
357
358
359
360
361
362
363
364
365
366
367
368
369
370
371
372
373
374
375
376
377
378
379
380
381
382
383
384
385
386
387
388
389
390
391
392
393
394
395
396
397
398
399
400
401
402
403
404
405
406
407
408
409
410
411
412
413
414
415
416
417
418
419
420
421
422
423
424
425
426
427
428
429
430
431
432
433
434
435
436
437
438
439
440
441
442
443
444
445
446
447
448
449
450
451
452
453
454
455
456
457
458
459
460
461
462
463
464
465
466
467
468
469
470
471
472
473
474
475
476
477
478
479
480
481
482
483
484
485
486
487
488
489
490
491
492
493
494
495
496
497
498
499
500
501
502
503
504
505
506
507
508
509
510
511
512
513
514
515
516
517
518
519
520
521
522
523
524
525
526
527
528
529
530
531
532
533
534
535
536
537
538
539
540
541
542
543
544
545
546
547
548
549
550
551
552
553
554
555
556
557
558
559
560
561
562
563
564
565
566
567
568
569
570
571
572
573
574
575
576
577
578
579
580
581
582
583
584
585
586
587
588
589
590
591
592
593
594
595
596
597
598
599
600
601
602
603
604
605
606
607
608
609
610
611
612
613
614
615
616
617
618
619
620
621
622
623
624
625
626
627
628
629
630
631
632
633
634
635
636
637
638
639
640
641
642
643
644
645
646
647
648
649
650
651
652
653
654
655
656
657
658
659
660
661
662
663
664
665
666
667
668
669
670
671
672
673
674
675
676
677
678
679
680
681
682
683
684
685
686
687
688
689
690
691
692
693
694
695
696
697
698
699
700
701
702
703
704
705
706
707
708
709
710
711
712
713
714
715
716
717
718
719
720
721
722
723
724
725
726
727
728
729
730
731
732
733
734
735
736
737
738
739
740
741
742
743
744
745
746
747
748
749
750
751
752
753
754
755
756
757
758
759
760
761
762
763
764
765
766
767
768
769
770
771
772
773
774
775
776
777
778
779
780
781
782
783
784
785
786
787
788
789
790
791
792
793
794
795
796
797
798
799
800
801
802
803
804
805
806
807
808
809
810
811
812
813
814
815
816
817
818
819
820
821
822
823
824
825
826
827
828
829
830
831
832
833
834
835
836
837
838
839
840
841
842
843
844
845
846
847
848
849
850
851
852
853
854
855
856
857
858
859
860
861
862
863
864
865
866
867
868
869
870
871
872
873
874
875
876
877
878
879
880
881
882
883
884
885
886
887
888
889
890
891
892
893
894
895
896
897
898
899
900
901
902
903
904
905
906
907
908
909
910
911
912
913
914
915
916
917
918
919
920
921
922
923
924
925
926
927
928
929
930
931
932
933
934
935
<!DOCTYPE html>
<html lang="" xml:lang="">
<head>
<title>Bonnes pratiques en bioinformatique : (essayer) d’aller vers plus de reproductibilité</title>
<meta charset="utf-8" />
<meta name="author" content="Valentin Loux - Cédric Midoux - Olivier Rué" />
<meta name="date" content="2021-03-08" />
<link href="slides_files/remark-css/default.css" rel="stylesheet" />
<link href="slides_files/remark-css/default-fonts.css" rel="stylesheet" />
<link rel="stylesheet" href="../css/styles.css" type="text/css" />
</head>
<body>
<textarea id="source">
class: center, middle, inverse, title-slide
# Bonnes pratiques en bioinformatique : (essayer) d’aller vers plus de reproductibilité
## DUBii - Module 5
### Valentin Loux - Cédric Midoux - Olivier Rué
### 2021-03-08
---
class: hide-logo
# Programme de l'après-midi
<!-- 14-17h -->
- Intro : Quelques généralités sur la reproductibilité et la démarche science ouverte <!-- 25 mn) -->
- Partie 1 : Organiser son espace de travail <!-- 5 mn -->
- Partie 2 : Versionner ses documents <!-- 45 mn -->
- TP
<!-- 15h45 Pause -->
- Partie 3 : Des langages à faible balisage pour faciliter la traçabilité et la prise de note <!-- 30 mn -->
- Partie 4 : Utilisation de documents computationels -- Notebook <!-- 45 mn -->
- TP
- Conclusion & aller + loin <!-- 15 mn -->
---
# Tout le monde a déjà eu cette expérience
--
.pull-left[
<div class="figure" style="text-align: center">
<img src="images/monnet-loux-plosone-2010.png" alt="Un article interessant" width="100%" />
<p class="caption">Un article interessant</p>
</div>
]
--
.pull-right[
<div class="figure" style="text-align: center">
<img src="images/monnet-loux-plosone-mat-met.png" alt="Un matériel et méthodes décevant" width="100%" />
<p class="caption">Un matériel et méthodes décevant</p>
</div>
]
--
---
# Crise de la reproductibilité
- Problème **général**, "Reproducibility Crisis"
- Remis en avant par les science sociales, notamment la psychologie
- Étendu à l'ensemble des disciplines scientifiques
Mais un problème qui n'est **pas nouveau** :
- Expériences de la pompe à vide au XVIIe siècle (von Guericke et Boyle)
<div class="figure" style="text-align: center">
<img src="images/Magdeburger-Halbkugeln.jpg" alt="Experiences des hemisphères de Magdebourg" width="40%" />
<p class="caption">Experiences des hemisphères de Magdebourg</p>
</div>
???
Au XVIIe, expérience de la pompe à vide par Otto van Guericke étaient basées sur des démonstrations spectaculaires, dans les cours d'Europe qui lui assuraient notoriété. L'expérience des hemisphères de Magdebourg qui consistait à essayer de faire séparer des hémisphères de cuivre dans lesquels il avait fait le vide, par des attelages de chevaux était très appréciée. Cela reposait sur un outillage et un savoir faire secret. Impossible à reproduire
Plus tard, Boyle, sur le même sujet, consignait tout par écrit et avait recours à des témoins de bonne réputation (des gentlemen, à la base de la Royal Society) pour certifier ses expériences. c'est le début des publication scientifiques.
---
# Et en bioinfo ?
Un problème vieux comme la bioinformatique :
- En 2009, moins de la moitié des expériences de transcriptomique parues dans Nature Genetics ont pu être reproduites
- Sur 50 articles citant BWA en 2011, 31 ne citent ni version, ni paramètres. 26 ne donnent pas accès aux données sous-jacentes
- Selon un sondage mené en 2016 auprès de plus de 1500 biologistes
- 70% ont déjà éprouvé des difficultés à reproduire une analyse ([Baker, 2016](https://doi.org/10.1038/533452a))
- [Ten Years Reproducibility Challenge](https://github.com/ReScience/ten-years) : êtes vous capables de refaire vos analyses d'il y a 10 ans ?
---
# Quelles sont les difficultés ?
- Problèmes **d'accès aux données** :
- le fameux "data available upon request"
- données brutes disponibles, mais méta-données inexistantes ou insuffisantes
- Problèmes **d'accès aux outils** :
- outils anciens ou obsolètes
- difficultés à installer
- Problèmes de **paramètrage de l'analyse**
- version des outil
- paramètres des outils
- enchaînement des outils
- Problème d'accès aux **ressources necessaires**
- calcul
- stockage
---
# Réplication ≠ Reproductibilité
- La réplication indépendante d'expériences est à la base de la méthode scientifique
- En complément de **réplication** indépendante ( expérimentation, échantillonnage, analyse, …), la **reproduction** d'analyse est indispensable à l'évaluation et à la compréhension de la démarche employée
- Il existe une ambiguïté en anglais entre réplication (*réplication*) et reproduction (*reproducibility*). Derrière la *reproducibility crisis* on mélange les deux :
- Impossibilité de répliquer des résultats de façon indépendante (psychologie, médecine, biologie…)
- Impossibilité de reproduire des analyses à partir des mêmes données de départ
- Chacun peut déjà, par la mise en place de pratiques simples et l'utilisation d'outils conviviaux, améliorer la reproductibilité de ses travaux
Source : ([Allard, 2018](https://laviedesidees.fr/La-crise-de-la-replicabilite.html))
<!-- https://laviedesidees.fr/La-crise-de-la-replicabilite.html -->
---
# En pratique, qu'est ce qu'être reproductible ?
<div class="figure" style="text-align: left">
<img src="images/reproducible-data-analysis.png" alt="https://github.com/karthik/rstudio2019/blob/master/reproducible-data-analysis.pdf" width="80%" />
<p class="caption">https://github.com/karthik/rstudio2019/blob/master/reproducible-data-analysis.pdf</p>
</div>
---
# En pratique, qu'est ce qu'être reproductible (2) ?
Avoir accès :
- aux pièces (les **données**)
- aux outils ( les **logiciels**, )
- au mode d'emploi : **paramètres**, **workflows d'analyse**
Mais aussi :
- à la description des pièces, de la façon dont elles ont été produites (**méta-données**)
- à la documentation technique (**choix techniques explicites**)
- au savoir faire du monteur (**formations**)
- Éventuellement à un atelier équipé pour le montage (**ressources informatiques**)
---
# FAIR : un pré-requis à la reproductibilité
.pull-left[
<div class="figure" style="text-align: left">
<img src="images/fair.png" alt="Findale Accessible Interoperable Reusable " width="50%" />
<p class="caption">Findale Accessible Interoperable Reusable </p>
</div>
]
.pull-right[
Principes autour des données au sens large :
- **F**acile à trouver : pour les humains et les ordinateurs :
- id unique et pérennes
- métadonnées riches
- **A**ccessibles à ** long terme**
- entrepôt "pérenne"
- licence d'utilisation explicite ( FAIR ≠ ouvert)
- **I**nteroperables : faciles à combiner avec d'autres jeux de données
- formats ouverts et documentés
- vocabulaire standardisé, ontologies (données et méta-données)
- **R**éutilisables :
- réutilisables par soi, par d'autres
- réutilisables par des machines
]
([Wilkinson, Dumontier, Aalbersberg, et al., 2016](https://doi.org/10.1038/sdata.2016.18))
---
# Le spectre de la reproductibilité
<div class="figure" style="text-align: center">
<img src="images/reproducibility-spectrum.png" alt="Spectre de la reproductibilité," width="100%" />
<p class="caption">Spectre de la reproductibilité,</p>
</div>
Source : ([Piazzi, Cerqueira, Manso, et al., 2018](#bib-piazzi))
---
# En pratique, que faire, quels outils utiliser ?
Aller de façon **pragmatique** vers une documentation accrue de ce que l'on fait (comment, pourquoi) et des données que l'on utilise et produit.
- Rendre accessible ses données à soit, aux partenaires, à tous) :
- Documenter collectivement ses pratiques de gestion de données (responsabilités, formats, cycle de vie, archivage…) dns un Data Management Plan qui doit être **vivant** et partagé.(Opidor)
- Dépôt internationaux (ENA, NCBI) pour les données specialisées
- DataVerse, Figshare, Zenodo ou autre pour les autres données
- Définir et fixer les versions des outils utilisés :
- Conda, Bioconda
- Singularity, Docker
- Machine Virtuelle
- Décrire son workflow d'analyse, le rendre portable :
- Galaxy
- Snakemake, Nextflow
- Gérer les versions de ses codes, les publier :
- git
- github / gitlab
- Tracer son analyse dans des documents computationnels partageables et réutilisables :
- Rmd
- Jupyter Notebooks
---
# Objectifs du TP
Décomplexifier les problème, se décomplexifier sur ses pratiques , désacraliser la reproductibilité !
Vous fournir des outils, des pistes pour rendre vos projets :
- transparents
- robustes
- réutilisables
- partageables
Bref, *plus* ouverts et reproductibles.
Parties pratiques sur la versionning des documents (Git et GitHub) et les documents computationnels.
---
class: inverse, center, middle
# Partie 1 : Organiser son espace de travail
---
# Partie 1 : Organiser son espace de travail
.pull-left[

Source : ([Noble, 2009](https://doi.org/10.1371/journal.pcbi.1000424))
]
.pull-right[
Séparer
- données
- code
- scripts
- résultats
- Avoir un copie de sauvegarde de ses données
- Mettre le repertoire et les fichiers de données en lecture seule
- Avoir une convention de nommage de ses fichiers
]
---
# Organiser son espace de travail (2)
<img src="images/Infographic_Data_Management_v02-02_KH.png" width="50%" style="display: block; margin: auto;" />
Source : [Twitter Kira Höffler](https://twitter.com/KiraHoeffler/status/1367804034413920259) | [Version PDF](images/Infographic_Data_Management_v02-02_KH.pdf)
---
class: inverse, center, middle
# Partie 2 : Versionner ses documents
---
<div class="figure" style="text-align: center">
<img src="images/phd101212s.gif" alt="Piled Higher and Deeper by Jorge Cham. phdcomics" width="40%" />
<p class="caption">Piled Higher and Deeper by Jorge Cham. phdcomics</p>
</div>
---
# Partie 2 : Versionner ses documents
.pull-left[
- Les documents évoluent, il est nécéssaire de suivre les versions
- On trace toutes les modifications faites
- On garde chaque version des documents du dossier de travail
- C’est un peu comme copier-coller son dossier de travail … mais en beaucoup plus précis et pratique !
- `git` est un standard dans la gestion des versions distribuée
]
.pull-right[

]
Voici un [aide mémoire git](https://github.github.com/training-kit/downloads/fr/github-git-cheat-sheet.pdf)
---
# Vocabulaire
.pull-left[
Quelques mots à connaitre :
- **Repository** / **Dépôt** = Dossier / Projet
- **Commit** = Enregistrement d’un ensemble de fichier à un instant T (= photo)
- **Branche** = Ensemble chaîné de commits, par défaut la branche principale s'appelle « main »
- **Git** : logiciel *open-souce* de gestion de version de document. Il est tout a fait possible d'utiliser git pour versionner ses documents sans GitHub.
- **Github** : site web permettant de centraliser en ligne ses dépôts git et facilitant la collaboration sur les projets.
]
.pull-right[
<img src="images/git-github.png" width="100%" style="display: block; margin: auto 0 auto auto;" />
]
---
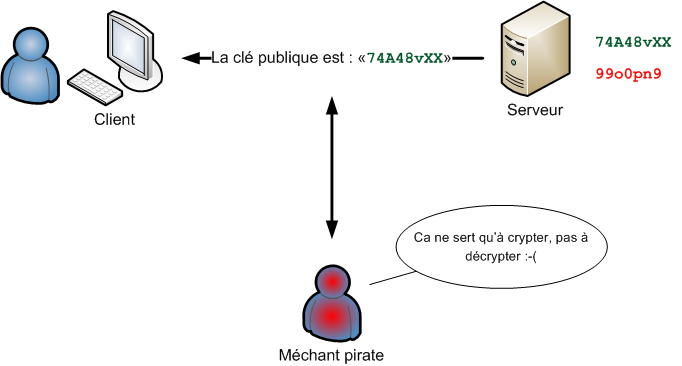
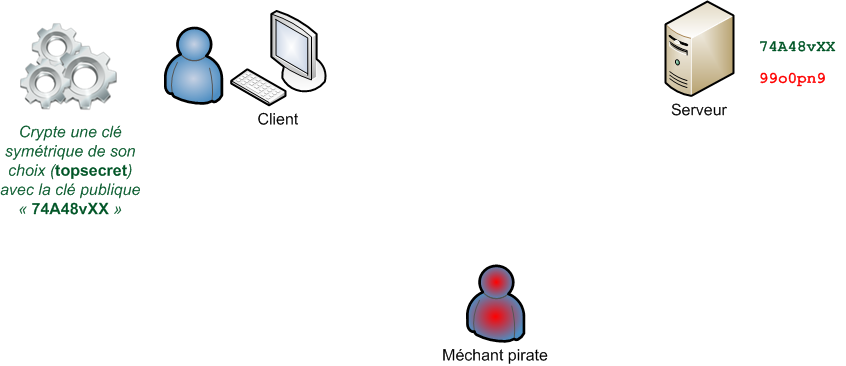
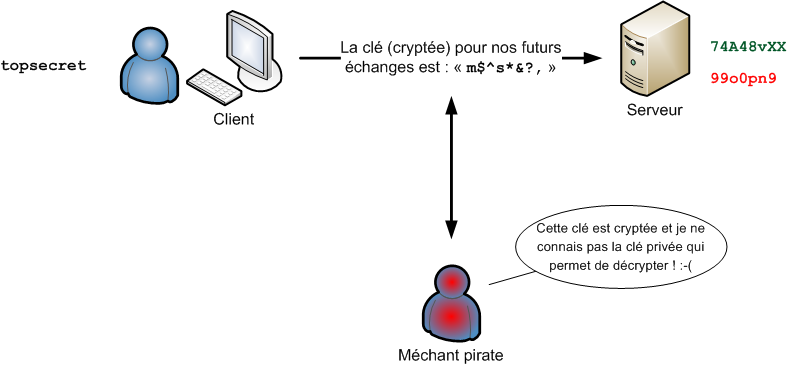
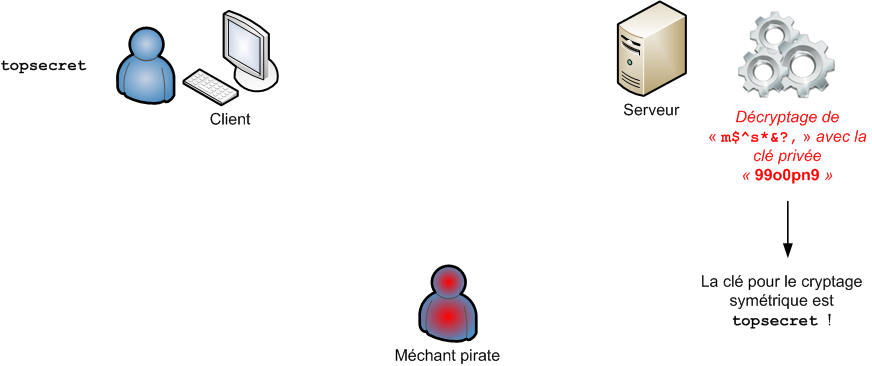
# Retours sur les clés SSH
* Protocole pour la sécurisation des transferts de données.
* Méthode de chiffrement asymétrique qui fonctionne avec une paire de clé :
* une clé *publique* qui sert à chiffrer (et que vous pouvez partager à l'extérieur)
* une clé *privée* qui sert à déchiffrer (et que vous gardez précieusement secrète)
* Permet d'établir un tunnel sécurisé entre deux machines
---
class: center, middle
---
class: center, middle
---
class: center, middle
---
class: center, middle
---
class: center, middle
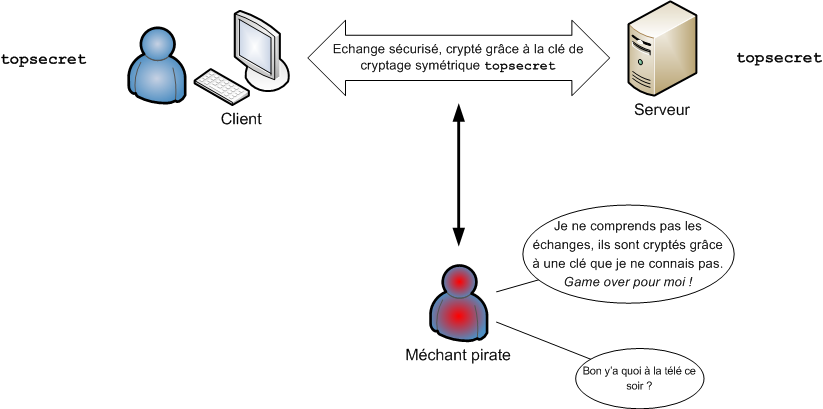
---
# Retours sur les clés SSH
* Protocole pour la sécurisation des transferts de données.
* Méthode de chiffrement asymétrique qui fonctionne avec une paire de clé :
* une clé *publique* qui sert à chiffrer (et que vous pouvez partager à l'extérieur)
* une clé *privée* qui sert à déchiffrer (et que vous gardez précieusement secrète : `chmod 400 ~/.ssh/id_rsa`)
* Permet d'établir un tunnel sécurisé entre deux machines
---
# Comment générer une clef, en pratique :
* Sous Linux, on utilise `ssh-keygen -t rsa` pour générer la paire de clé
* Puis `ssh-copy-id -i id_rsa.pub <login>@<server>` si on veut envoyer la clé publique sur un serveur distant
* Maintenant on peut se connecter de manière sécurisée via `ssh <login>@<server>` sans renseigner de clé
* Pour **GitHub**, **Rstudio** on renseigne les clés publiques via l'interface graphique
---
class: center, middle
# TP : générer sa clef ssh
---
---
class: center, middle
# TP : Utilisation de Git
---
# Git : En résumé
- `git clone` : cloner un dépot distant
- `git init` : initialiser le versionning sur un dépot local
- `git commit` : enregister l'état d'un dépôt
- `git status` : afficher l'état des documents du dépôt
- `git diff` : comparer l'état actuel au dernier commit, ou deux commits entre eux ou deux branches
- `git pull` : récupérer les commits distants
- `git push` : envoyer les commits locaux
et bien d'autres encore (`blame`, `revert`,…)
[CheatSheet](https://github.github.com/training-kit/downloads/github-git-cheat-sheet.pdf)
---
# Pour apprendre progressivement et/ou se simplifier la vie
Utiliser une interface conviviale :
- Github Desktop (Windows et macOS)
- Les intégration aux différents IDE (RStudio and co)
.center[
.pull-left[]
.pull-right[]
]
---
<div class="figure" style="text-align: center">
<img src="images/github_desktop_1.png" alt="Cloner un dépot" width="80%" />
<p class="caption">Cloner un dépot</p>
</div>
---
<div class="figure" style="text-align: center">
<img src="images/github_desktop_2.png" alt="Suivre les changements" width="80%" />
<p class="caption">Suivre les changements</p>
</div>
---
<div class="figure" style="text-align: center">
<img src="images/github_desktop_3.png" alt="Parcourir l'historique des modifications" width="80%" />
<p class="caption">Parcourir l'historique des modifications</p>
</div>
---
<div class="figure" style="text-align: center">
<img src="images/github_desktop_4.png" alt="Intéragir avec le Repository" width="80%" />
<p class="caption">Intéragir avec le Repository</p>
</div>
---
class: center, middle

---
class: inverse, center, middle
# Partie 3 : Des langages à faible balisage pour faciliter la traçabilité et la prise de note
---
# Partie 3 : Des langages à faible balisage pour faciliter la traçabilité et la prise de note
*Comment mettre en forme et structurer simplement un document texte ?*
--
&rarr; avec un balisage faible tel que *Markdown*
--
.pull-left[
- Permet :
- Organiser les titres de sections
- Italique / gras / souligné
- Générer des listes
- Ajouter des tableaux
- Insertion d'image et de blocs de code
- Texte codé en UTF-8 (assure une pérennité, lisibilité et portabilité) facilement versionnable.
- Les langages de balisage permettent de mettre en forme convenablement le fichier pour un meilleur confort de lecture.
]
--
.pull-right[
_Exemple illustrant la simplification du balisage :_
- HTML
```bash
<ul>
<li>item1</li>
<li>item2</li>
</ul>
```
- Markdown
```bash
- item1
- item2
```
]
---
# Partie 3 : Markdown - exemples de mise en forme
````
# Titre H1
## Sous-titre H2
### Sous-titre H3
*italique*, **gras** et `code`
> Citations
```
bloc de code
```
* liste
* item
* item
[lien](https://fr.wikipedia.org)

#lien vers une image en ligne ou dans l'espace de travail
````
Gardez ce [mémo](https://guides.github.com/pdfs/markdown-cheatsheet-online.pdf) à porté de main !
---
class: inverse, center, middle
# Partie 4 : Documents computationels - Notebook
---
# Partie 4 : Documents computationels - Notebook
- Il faut se donner les moyens pour qu’autrui puisse inspecter nos analyses
- Expliciter pour augmenter les chances de trouver les erreurs et de les éliminer
- Inspecter pour justifier et comprendre
- Refaire pour vérifier, corriger et réutiliser
.center[]
---
# Partie 4 : Documents computationels - Notebook
- Regrouper dans *un unique document*:
- Les informations, le code, calculs et les résultats
- Pour assurer leur cohérence et améliorer la traçabilité.
- Exportable (ex : html) pour une meilleure portabilité et lisibilité.
<img src="https://www.researchgate.net/profile/Dario_Righelli/publication/306538473/figure/fig2/AS:391153263366144@1470269613039/This-figure-explains-the-basic-idea-of-Reproducible-Research-The-raw-data-needs-to-be_W640.jpg" width="50%" style="display: block; margin: auto;" />
([Russo, Righelli, and Angelini, 2016](https://doi.org/10.1007/978-3-319-44332-4_19))
Encore un joli [mémo](https://www.rstudio.org/links/r_markdown_cheat_sheet) pour R markdown.
Si vous travaillez principalement avec Python, il est possible de faire des notebook avec Jupyter.
---
class: center, middle
# TP : Documents computationels
---
# Documents computationels : En résumé
**Dans un unique document on a** :
- Une entête générale
- Du texte, mis en forme avec markdown
- Du code R ou Python (ou autre) dans des chunks
- Des résultats, plots et outputs
* Ce document est versionné
* Il est dans un format texte dont la lisibilité est assurée au cours du temps
* On peut l'exporter en HTML et le rendre accessible via les GitHub Pages.
** &rarr; Expliciter pour augmenter les chances de trouver les erreurs et de les éliminer **
- Inspecter pour justifier et comprendre
- Refaire pour vérifier, corriger et réutiliser
---
# Conclusion :
La reproductibilité, comme la "FAIRisation" sont des processus.
Des bonnes pratiques appuyées par des outils qui les facilitent
- Organiser ses analyses
- Décrire correctement ses données et ses processus d'analyse (PGD, FAIR)
- Tracer ses analyses à l'aide de documents computationels (Rmd, Jupyter Notebooks, …) :
- Transparents
- Accessibles
- Partageables
- Versionner ses documents computationnels (GitHub, GitLab, …)
- Traçabilité
- Accessibilité
---
# Aller plus loin - Travailler en commun avec Git et Github :
.center[]
- Branche : version parallèle à la version principale
- Pull Request : demande de fusion des modifications d'une brache vers la branche principale
[Un exemple de PR](https://github.com/galaxyproject/training-material/pull/1354)
---
# Pour aller + loin - Fixer et partager son environnement :
.pull-left[
- Conda et Bioconda
- gestion des dépendances, versions
- Possibilité de créer un environnement par analyse
- Exporter son environnement dans un fichier `env.yml` et le versionner
- `conda env export > environment.yml`
- Containers, machines virtuelles
- Docker, Singularity, VM virtualbox
- Pour les outils non "conda-isables", les environnements complexes
- Les images Singularity sont déployables sur les infrastructures type IFB et s'éxecutent "presque" comme un executable
]
.pull-right[
.center[]
.center[]
]
---
# Pour aller plus loin - Fixer et partager son environnement (2)
.center[]
([Grüning, Chilton, Köster, et al., 2018](https://doi.org/10.1016/j.cels.2018.03.014))
---
# Pour aller + loin - Gestionnaires de workflows
SnakeMake, NextFlow pour :
- Définir de façon "simple" et modulaire des workflows d'analyse :
- Parallelisables : les étapes indépendantes peuvent être jouées en parallèle.
- Qui assurent la reprise sur erreur : si on refait une analyse, change un paramètre, seul ce qui doit être rejoué est relancé.
- Portables : un même script peut être joué en local, sur des clusters différents en changeant le fichier de configuration.
- Partageables : un fichier texte versionné
- Peut gérer pour vous le versionning et l'installation des outils avec Conda
---
# Pour aller + loin - exemple de Snakefile
**Bash**
```
for sample in `ls *.fastq.gz` do
fastqc ${sample}
done
```
--
**Snakefile**
```
SAMPLES, = glob_wildcards("./{smp}.fastq.gz")
rule final:
input:expand("fastqc/{smp}/{smp}_fastqc.zip",smp=SAMPLES)
rule fastqc:
input: "{smp}.fastq.gz"
output: "fastqc/{smp}/{smp}_fastqc.zip"
message: """Quality check"""
shell: """fastqc {input} --outdir fastqc/{wildcards.smp}"""
```
---
# Pour aller + loin - FAIRifier ses données
Dépots dans les dépôts publics :
- Dans les dépôts thématiques internationaux (européens !)
- données brutes
- données analysées
- /!\ méta-données
- dans les dépots généralistes (dataverse , figshare, …)
- fichiers tabulés; "autres" données. ce qu'on mettrait en suypplementary material.
- (éventuels) leins vers les fichiers de données
- Publier un data-paper ?
---
# Pour aller + loin :
.pull-left[
- jusqu'où aller dans la reproductibilité ?
- Mat et Met électroniques :
- Galaxy Pages
- Gigascience, GigaDB :
- "GigaScience aims to revolutionize publishing by promoting reproducibility of analyses and data dissemination, organization, understanding, and use. "
]
.pull-right[
<div class="figure" style="text-align: center">
<img src="images/galaxy-pages.png" alt="Live Mat et Met https://usegalaxy.org/u/aun1/p/windshield-splatter" width="80%" />
<p class="caption">Live Mat et Met https://usegalaxy.org/u/aun1/p/windshield-splatter</p>
</div>
Au final, toujours se poser la question du rapport coût / bénéfice.
]
---
# Ressources
- [FUN MOOC Recherche Reproductible](https://www.fun-mooc.fr/courses/course-v1:inria+41016+self-paced/about)
- [FAIR Bioinfo](https://github.com/thomasdenecker/FAIR_Bioinfo)
- [Cours Git et Github](https://guides.github.com)
- [Github pages](https://pages.github.com)
- [Rmd the definitive Guide](https://bookdown.org/yihui/rmarkdown/)
- [Snakemake](https://snakemake.readthedocs.io/en/stable/)
- [NextFlow](https://www.nextflow.io) et [nf-core](https://nf-co.re)
-
- Les mémo présentés dans ce cours :
- [markdown](https://guides.github.com/pdfs/markdown-cheatsheet-online.pdf)
- [git](https://github.github.com/training-kit/downloads/fr/github-git-cheat-sheet.pdf)
- [R markdown](https://www.rstudio.org/links/r_markdown_cheat_sheet)
---
# References
<a name=bib-allard></a>[Allard, A.](#cite-allard) (2018). _La crise de la réplicabilité_. URL:
[https://laviedesidees.fr/La-crise-de-la-replicabilite.html](https://laviedesidees.fr/La-crise-de-la-replicabilite.html).
<a name=bib-Baker2016></a>[Baker, M.](#cite-Baker2016) (2016). "1, 500 scientists lift the lid on
reproducibility". In: _Nature_ 533.7604, pp. 452-454. DOI: [10.1038/533452a](https://doi.org/10.1038%2F533452a).
URL: [https://doi.org/10.1038/533452a](https://doi.org/10.1038/533452a).
<a name=bib-Bjorn2018></a>[Grüning, B., J. Chilton, J. Köster, et al.](#cite-Bjorn2018) (2018). "Practical
Computational Reproducibility in the Life Sciences". In: _Cell Systems_ 6.6, pp. 631-635. DOI:
[10.1016/j.cels.2018.03.014](https://doi.org/10.1016%2Fj.cels.2018.03.014). URL:
[https://doi.org/10.1016/j.cels.2018.03.014](https://doi.org/10.1016/j.cels.2018.03.014).
<a name=bib-noble></a>[Noble, W. S.](#cite-noble) (2009). "A Quick Guide to Organizing Computational Biology
Projects". In: _PLOS Computational Biology_ 5.7, pp. 1-5. DOI:
[10.1371/journal.pcbi.1000424](https://doi.org/10.1371%2Fjournal.pcbi.1000424). URL:
[https://doi.org/10.1371/journal.pcbi.1000424](https://doi.org/10.1371/journal.pcbi.1000424).
<a name=bib-piazzi></a>[Piazzi, A. C., A. S. Cerqueira, L. R. Manso, et al.](#cite-piazzi) (2018). "Reproducible
research platform for electric power quality algorithms" , pp. 1-6.
<a name=bib-russo></a>[Russo, F., D. Righelli, and C. Angelini](#cite-russo) (2016). "Advantages and Limits in
the Adoption of Reproducible Research and R-Tools for the Analysis of Omic Data". In: Vol. 9874. , pp. 245-258.
DOI: [10.1007/978-3-319-44332-4_19](https://doi.org/10.1007%2F978-3-319-44332-4_19).
<a name=bib-Wilkinson2016></a>[Wilkinson, M. D., M. Dumontier, I. J. Aalbersberg, et al.](#cite-Wilkinson2016)
(2016). "The FAIR Guiding Principles for scientific data management and stewardship". In: _Scientific Data_ 3.1.
DOI: [10.1038/sdata.2016.18](https://doi.org/10.1038%2Fsdata.2016.18). URL:
[https://doi.org/10.1038/sdata.2016.18](https://doi.org/10.1038/sdata.2016.18).
<a name=bib-Wilson></a>[Wilson, G., J. Bryan, K. Cranston, et al.](#cite-Wilson) (2017). "Good enough practices
in scientific computing". In: _PLOS Computational Biology_ 13, pp. 1-20. DOI:
[10.1371/journal.pcbi.1005510](https://doi.org/10.1371%2Fjournal.pcbi.1005510). URL:
[https://doi.org/10.1371/journal.pcbi.1005510](https://doi.org/10.1371/journal.pcbi.1005510).
</textarea>
<style data-target="print-only">@media screen {.remark-slide-container{display:block;}.remark-slide-scaler{box-shadow:none;}}</style>
<script src="https://remarkjs.com/downloads/remark-latest.min.js"></script>
<script>var slideshow = remark.create({
"ratio": "16:9"
});
if (window.HTMLWidgets) slideshow.on('afterShowSlide', function (slide) {
window.dispatchEvent(new Event('resize'));
});
(function(d) {
var s = d.createElement("style"), r = d.querySelector(".remark-slide-scaler");
if (!r) return;
s.type = "text/css"; s.innerHTML = "@page {size: " + r.style.width + " " + r.style.height +"; }";
d.head.appendChild(s);
})(document);
(function(d) {
var el = d.getElementsByClassName("remark-slides-area");
if (!el) return;
var slide, slides = slideshow.getSlides(), els = el[0].children;
for (var i = 1; i < slides.length; i++) {
slide = slides[i];
if (slide.properties.continued === "true" || slide.properties.count === "false") {
els[i - 1].className += ' has-continuation';
}
}
var s = d.createElement("style");
s.type = "text/css"; s.innerHTML = "@media print { .has-continuation { display: none; } }";
d.head.appendChild(s);
})(document);
// delete the temporary CSS (for displaying all slides initially) when the user
// starts to view slides
(function() {
var deleted = false;
slideshow.on('beforeShowSlide', function(slide) {
if (deleted) return;
var sheets = document.styleSheets, node;
for (var i = 0; i < sheets.length; i++) {
node = sheets[i].ownerNode;
if (node.dataset["target"] !== "print-only") continue;
node.parentNode.removeChild(node);
}
deleted = true;
});
})();
(function() {
"use strict"
// Replace <script> tags in slides area to make them executable
var scripts = document.querySelectorAll(
'.remark-slides-area .remark-slide-container script'
);
if (!scripts.length) return;
for (var i = 0; i < scripts.length; i++) {
var s = document.createElement('script');
var code = document.createTextNode(scripts[i].textContent);
s.appendChild(code);
var scriptAttrs = scripts[i].attributes;
for (var j = 0; j < scriptAttrs.length; j++) {
s.setAttribute(scriptAttrs[j].name, scriptAttrs[j].value);
}
scripts[i].parentElement.replaceChild(s, scripts[i]);
}
})();
(function() {
var links = document.getElementsByTagName('a');
for (var i = 0; i < links.length; i++) {
if (/^(https?:)?\/\//.test(links[i].getAttribute('href'))) {
links[i].target = '_blank';
}
}
})();</script>
<style>
.logo-inra {
background-image: url(images/logos/Logo-INRAE_Transparent.png);
background-size: contain;
background-repeat: no-repeat;
position: fixed;
bottom: -4em;
width: 53px;
height: 110px;
z-index: 0;
margin-bottom: -10px;
left:25em;
}
.logo-migale {
background-image: url(images/logos/migale-orange.png);
background-size: contain;
background-repeat: no-repeat;
position: fixed;
bottom: -4em;
width: 85px;
left: 7em;
height: 116px;
z-index: 0;
margin-bottom: -10px;
left:28em;
}
.logo-univ {
background-image: url(../images/logo_UParis.png);
background-size: contain;
background-repeat: no-repeat;
position: fixed;
width: 205px;
left: 7em;
height: 60px;
z-index: 0;
left: 20em;
}
.logo-ifb {
background-image: url(../images/logo-ifb_small.png);
background-size: contain;
background-repeat: no-repeat;
position: fixed;
width: 118px;
left: 7em;
height: 100%;
z-index: 0;
left: 34em;
}
.logo-inrae {
background-image: url(../images/inrae.png);
background-size: contain;
background-repeat: no-repeat;
position: fixed;
width: 118px;
left: 7em;
height: 100%;
z-index: 0;
left: 16em;
}
.logo-migale {
background-image: url(../images/migale-orange.png);
background-size: contain;
background-repeat: no-repeat;
position: fixed;
width: 118px;
left: 7em;
height: 100%;
z-index: 0;
left: 18em;
}
</style>
<script>
document
.querySelectorAll(
'.remark-slide-content' +
'.title-slide' +
// add additional classes to exclude here, e.g.
// ':not(.inverse)' +
':not(.hide-logo)'
)
.forEach(el => {
//el.innerHTML += '<div class="logo-inra"></div>';
//el.innerHTML += '<div class="logo-migale"></div>';
el.innerHTML += '<p style="text-align: center;"> \
<img src="../images/logo-ifb_small.png" height="60px"></img> \
<img src="../images/logo_UParis.png" height="60px"></img></p>';
el.innerHTML += '<p style="text-align: center;"><img src="../images/migale-orange.png" height="40px"></img> \
<img src="../images/inrae.png" height="35px"></img></p><p style="text-align: center;"><img src="../images/licence.png" height="40px"></img></p>';
});
</script>
<script>
slideshow._releaseMath = function(el) {
var i, text, code, codes = el.getElementsByTagName('code');
for (i = 0; i < codes.length;) {
code = codes[i];
if (code.parentNode.tagName !== 'PRE' && code.childElementCount === 0) {
text = code.textContent;
if (/^\\\((.|\s)+\\\)$/.test(text) || /^\\\[(.|\s)+\\\]$/.test(text) ||
/^\$\$(.|\s)+\$\$$/.test(text) ||
/^\\begin\{([^}]+)\}(.|\s)+\\end\{[^}]+\}$/.test(text)) {
code.outerHTML = code.innerHTML; // remove <code></code>
continue;
}
}
i++;
}
};
slideshow._releaseMath(document);
</script>
<!-- dynamically load mathjax for compatibility with self-contained -->
<script>
(function () {
var script = document.createElement('script');
script.type = 'text/javascript';
script.src = 'https://mathjax.rstudio.com/latest/MathJax.js?config=TeX-MML-AM_CHTML';
if (location.protocol !== 'file:' && /^https?:/.test(script.src))
script.src = script.src.replace(/^https?:/, '');
document.getElementsByTagName('head')[0].appendChild(script);
})();
</script>
</body>
</html>